
KeyWord: バイオインフォマティクス,タンパク質,糖鎖修飾,ニューラルネットワーク,SVM
1、研究背景・目的
2003年ヒトゲノム計画が完了し、ヒトのゲノムの全塩基配列が解析されました。ゲノムはタンパク質を発現することにより役割(機能)を果たします。
現在はゲノムから発現したタンパク質の構造と機能の解析をおこなうプロテオーム解析が注目を浴びています。
ヒトのタンパク質数はタンパク質をコードしている遺伝子の数よりもずっと多いことが判明し、このようなタンパク質の多様性は翻訳後修飾からきています。 本研究ではその翻訳後修飾のうちのO-型糖鎖修飾を対象としています。タンパク質の過半数が糖鎖修飾を受けることが判明しており、 修飾異常があったときにアルツハイマー病や癌などの重大な疾患を生じさせる原因のひとつになっているといわれています。
糖鎖修飾には主にN-型糖鎖修飾とO-型糖鎖修飾があり、 N-型糖鎖修飾についてはコンセンサス配列(どのようなアミノ酸配列のときに修飾を受けるか)が判明していますがO-型糖鎖修飾については
セリン・トレオニン残基に修飾を受けることは判明していますが、そのコンセンサス配列の有無・修飾機構が未解明であります。
O-型糖鎖修飾部位の予測をおこなうことにより、修飾に関与する要因や条件を明らかにし、生命現象の解明や創薬につなげることを目的としています。
N-型糖鎖修飾についてはコンセンサス配列(どのようなアミノ酸配列のときに修飾を受けるか)が判明していますがO-型糖鎖修飾については
セリン・トレオニン残基に修飾を受けることは判明していますが、そのコンセンサス配列の有無・修飾機構が未解明であります。
O-型糖鎖修飾部位の予測をおこなうことにより、修飾に関与する要因や条件を明らかにし、生命現象の解明や創薬につなげることを目的としています。
ヒトのタンパク質数はタンパク質をコードしている遺伝子の数よりもずっと多いことが判明し、このようなタンパク質の多様性は翻訳後修飾からきています。 本研究ではその翻訳後修飾のうちのO-型糖鎖修飾を対象としています。タンパク質の過半数が糖鎖修飾を受けることが判明しており、 修飾異常があったときにアルツハイマー病や癌などの重大な疾患を生じさせる原因のひとつになっているといわれています。
糖鎖修飾には主にN-型糖鎖修飾とO-型糖鎖修飾があり、
 N-型糖鎖修飾についてはコンセンサス配列(どのようなアミノ酸配列のときに修飾を受けるか)が判明していますがO-型糖鎖修飾については
セリン・トレオニン残基に修飾を受けることは判明していますが、そのコンセンサス配列の有無・修飾機構が未解明であります。
O-型糖鎖修飾部位の予測をおこなうことにより、修飾に関与する要因や条件を明らかにし、生命現象の解明や創薬につなげることを目的としています。
N-型糖鎖修飾についてはコンセンサス配列(どのようなアミノ酸配列のときに修飾を受けるか)が判明していますがO-型糖鎖修飾については
セリン・トレオニン残基に修飾を受けることは判明していますが、そのコンセンサス配列の有無・修飾機構が未解明であります。
O-型糖鎖修飾部位の予測をおこなうことにより、修飾に関与する要因や条件を明らかにし、生命現象の解明や創薬につなげることを目的としています。
2、研究手法
世界最大のタンパク質データベースであるUniProtよりO-型糖鎖修飾報告のある哺乳類タンパク質のアミノ酸配列情報を取得します。
取得したタンパク質のアミノ酸配列について
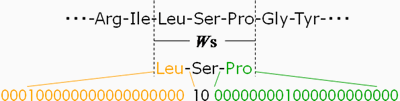 対象部位(セリン・トレオニン)を中心に一定幅(ウィンドウ幅Ws)で切り出し、
符号化を行い、2値(0・1)に変換します(左図)。
変換した値を階層型ニューラルネットワークやSupport Vector Machine(SVM)などの識別機を用いて修飾部位の予測をおこないます。
対象部位(セリン・トレオニン)を中心に一定幅(ウィンドウ幅Ws)で切り出し、
符号化を行い、2値(0・1)に変換します(左図)。
変換した値を階層型ニューラルネットワークやSupport Vector Machine(SVM)などの識別機を用いて修飾部位の予測をおこないます。
 対象部位(セリン・トレオニン)を中心に一定幅(ウィンドウ幅Ws)で切り出し、
符号化を行い、2値(0・1)に変換します(左図)。
変換した値を階層型ニューラルネットワークやSupport Vector Machine(SVM)などの識別機を用いて修飾部位の予測をおこないます。
対象部位(セリン・トレオニン)を中心に一定幅(ウィンドウ幅Ws)で切り出し、
符号化を行い、2値(0・1)に変換します(左図)。
変換した値を階層型ニューラルネットワークやSupport Vector Machine(SVM)などの識別機を用いて修飾部位の予測をおこないます。

